Ya sea un gusano, un ser humano o una ballena azul, toda la vida multicelular comienza como un huevo unicelular.
De esta celda solitaria emerge la galaxia de otros necesarios para construir un organismo, con cada célula nueva desarrollándose en el lugar correcto en el momento adecuado para llevar a cabo una función precisa en coordinación con sus vecinos.
Esta hazaña es una de las más notables en el mundo natural, y a pesar de décadas de estudio, una comprensión completa del proceso ha eludido a los biólogos.
Obtenga más noticias de HMS aquí
Ahora, en tres estudios publicados en Science , la Harvard Medical School y los investigadores de la Universidad de Harvard informan cómo han perfilado sistemáticamente cada célula en el desarrollo de embriones de pez cebra y rana para establecer una hoja de ruta que revela cómo una célula construye un organismo completo.
"Es casi como pasar de ver algunas estrellas a ver todo el universo" - Alexander Schier
Utilizando la tecnología de secuenciación de células individuales, los equipos de investigación rastrearon los destinos de las células individuales durante las primeras 24 horas de la vida de un embrión. Sus análisis revelan el panorama completo de los genes que se activan o desactivan, y cuándo, a medida que las células embrionarias pasan a nuevos estados y tipos de células.
En conjunto, los hallazgos representan un catálogo de "recetas" genéticas para generar diferentes tipos de células en dos especies modelo importantes y proporcionan un recurso sin precedentes para el estudio de la biología y la enfermedad del desarrollo.
"Con la secuenciación de células individuales, podemos, en un día de trabajo, recapitular décadas de investigación minuciosa sobre las decisiones que toman las células en las etapas más tempranas de la vida", dijo Allon Klein , profesor asistente de HMS de biología de sistemas y coautor de dos autores de los tres estudios de Ciencias .
Biomédicamente, estos recursos básicos para la forma en que se desarrollan los organismos son tan importantes como tener recursos de referencia para sus genomas, dijeron los investigadores.
"Con los enfoques que hemos desarrollado, estamos trazando un mapa de lo que creemos que será el futuro de la biología del desarrollo a medida que se transforma en una ciencia cuantitativa e impulsada por los grandes datos", dijo Klein.
Además de arrojar nueva luz sobre las primeras etapas de la vida, el trabajo podría abrir la puerta a una nueva comprensión de una serie de enfermedades, dijo Alexander Schier , profesor de Ciencias Biológicas Moleculares y Celulares Leo Erikson en Harvard, y su corresponsal. autor del tercer estudio.
"Prevemos que cualquier proceso biológico complejo en el que las células cambien la expresión génica a lo largo del tiempo se puede reconstruir utilizando este enfoque", dijo Schier. "No solo el desarrollo de embriones sino también el desarrollo del cáncer o la degeneración cerebral".

La secuenciación de células individuales permite a los investigadores estudiar la expresión de genes en miles de células, una célula a la vez. Video: Klein lab
Cada célula en un embrión en desarrollo lleva dentro una copia del genoma completo del organismo. Al igual que los trabajadores de la construcción que utilizan solo la parte relevante de un plan para establecer las bases de un edificio, las células deben expresar los genes necesarios en el momento apropiado para que el embrión se desarrolle correctamente.
En sus estudios, Klein colaboró con los coautores Marc Kirschner , el profesor de biología de sistemas de la HMS John Franklin Enders, Sean Megason , profesor asociado de HMS de biología de sistemas y colegas para analizar este proceso en el pez cebra y la rana occidental ( Xenopus tropicalis) ) embriones, dos de las especies modelo mejor estudiadas en biología.
Los investigadores aprovecharon el poder de InDrops , una tecnología de secuenciación de células individuales desarrollada en HMS por Klein, Kirschner y sus colegas, para capturar los datos de expresión génica de cada célula del embrión, una célula a la vez. Los equipos colectivamente perfilaron más de 200,000 células en múltiples puntos de tiempo durante 24 horas para ambas especies.
"Con estos conjuntos de datos, si alguien quiere hacer un tipo de celda específico, ahora tienen la receta" - Allon Klein
Para mapear el linaje de prácticamente cada célula a medida que se desarrolla un embrión, junto con la secuencia precisa de eventos de expresión genética que marcan nuevos estados y tipos de células, los equipos desarrollaron nuevas técnicas experimentales y computacionales, incluida la introducción de códigos de barras artificiales de ADN para rastrear el relaciones de linaje entre células, llamadas TracerSeq.
"Entender cómo se produce un organismo requiere saber qué genes se activan o desactivan a medida que las células toman decisiones sobre el destino, no solo la secuencia estática de un genoma", dijo Megason. "Este es el primer enfoque tecnológico que nos ha permitido abordar de forma sistemática y cuantitativa esta cuestión".
En el estudio codirigido por Schier, el equipo de investigación utilizó Drop-Seq, una tecnología de secuenciación unicelular desarrollada por investigadores de HMS y el Broad Institute of MIT y Harvard, para estudiar embriones de pez cebra durante 12 horas a alta resolución. Trabajando en equipo con Aviv Regev , miembro central de Broad, Schier y sus colegas reconstruyeron las trayectorias celulares a través de un método computacional que denominaron URD, después de la figura mitológica nórdica que decide todos los destinos.
Schier y sus colegas analizaron más de 38,000 células, y desarrollaron un "árbol genealógico" celular que reveló cómo la expresión génica en 25 tipos de células cambió a medida que se especializan. Al combinar esos datos con la inferencia espacial, el equipo también pudo reconstruir los orígenes espaciales de los distintos tipos de células en el embrión de pez cebra.

En ambas especies, los hallazgos de los equipos reflejaron gran parte de lo que se sabía anteriormente sobre la progresión del desarrollo embrionario, un resultado que subrayó el poder de los nuevos enfoques. Pero los análisis no tuvieron precedentes al revelar con detalles completos las cascadas de eventos que llevan a las células desde los estados progenitores tempranos o "generalistas" a estados más especializados con funciones estrechamente definidas.
Los equipos identificaron detalles difíciles de detectar como los tipos y subtipos de células raras y vincularon patrones de expresión génica nuevos y altamente específicos con diferentes linajes celulares. En varios casos, encontraron tipos de células emergentes mucho antes de lo que se pensaba.
"La belleza de trabajar en un organismo es que esto es todo. Diez, 20 años a partir de ahora, todavía podemos estar seguros de que el pez cebra y las ranas se desarrollarán de acuerdo con los mismos patrones "- Allon Klein
Para los científicos que se esfuerzan por responder preguntas sobre enfermedades humanas, estos datos podrían ser poderosamente esclarecedores. En la medicina regenerativa, por ejemplo, los investigadores han apuntado durante décadas a manipular las células madre hacia destinos específicos con el objetivo de reemplazar células, tejidos u órganos defectuosos por células funcionales. Los detalles recién recopilados sobre la secuencia de cambios en la expresión génica que precipitan la aparición de tipos celulares específicos pueden impulsar aún más estos esfuerzos.
"Con estos conjuntos de datos, si alguien quiere hacer un tipo de célula específico, ahora tienen la receta para los pasos que tomaron esas células cuando se formaron en el embrión", dijo Klein. "En algún sentido, hemos establecido una referencia de referencia para el desarrollo real de los procesos de diferenciación complejos en embriones, y establecemos un ejemplo de cómo reconstruir sistemáticamente este tipo de procesos".
Cuando se combina con uno de los conceptos básicos en la investigación biológica -la idea de alterar un sistema para estudiar lo que sucede-, la secuenciación de células individuales puede arrojar ideas difíciles de alcanzar antes, dijo Klein.
Como una prueba de principio, Klein, Megason y sus colegas utilizaron el sistema de edición de genes CRISPR / Cas9 para crear peces cebra con una forma mutante de cordina , un gen involucrado en la determinación de la orientación de un embrión en desarrollo. Schier y sus colegas adoptaron un enfoque similar al perfilar el pez cebra con una mutación en un gen diferente que se conoce como cabeza de alfiler de un solo ojo .
Cuando se analizaron con secuenciación de células individuales, los equipos confirmaron descripciones previamente conocidas de chordin y mutantes de cabeza de alfiler de un solo ojo , y pudieron describir en detalle o incluso predecir los efectos de estas mutaciones en células en desarrollo y tejidos nacientes en todo el embrión.
Inesperadamente, los grupos encontraron de forma independiente que a nivel de célula única, la expresión del gen era la misma en mutantes y en el tipo salvaje, a pesar de la pérdida de una ruta de señalización esencial. Las proporciones de diferentes tipos de células, sin embargo, cambiaron.
"Este trabajo solo fue posible gracias a tecnologías recientes que nos permiten analizar la expresión de genes en miles de células individuales", dijo Schier. "Ahora la escala es mucho más grande, por lo que podemos reconstruir la trayectoria de casi todas las células y todos los genes durante la embriogénesis. Es casi como pasar de ver algunas estrellas a ver todo el universo ".
Los equipos de investigación también demostraron cómo se pueden extraer estos datos para responder a preguntas fundamentales de larga data en biología.
Cuando Klein, Kirschner, Megason y sus colegas compararon paisajes de estado celular entre el pez cebra y los embriones de rana, observaron similitudes en su mayoría. Pero sus análisis también revelaron numerosas sorpresas. Una de tales observaciones fue que los genes que marcan los estados celulares en una especie a menudo son marcadores genéticos deficientes para el mismo estado celular en las otras especies.
En varios casos, descubrieron que la secuencia de ADN de un gen y la estructura de la proteína que codifica podrían ser casi idénticas entre las especies, pero tienen patrones de expresión muy diferentes.
"Esto realmente nos sorprendió, porque va en contra de toda la intuición que teníamos sobre el desarrollo y la biología", dijo Klein. "Fue una observación realmente incómoda. Desafía directamente nuestra idea de lo que significa ser un cierto "tipo de célula".
La razón por la cual estas diferencias no fueron detectadas antes, los investigadores sostienen la hipótesis, es que los análisis computacionales "prestan atención" a los datos de una manera fundamentalmente diferente de cómo los humanos lo hacen.
"Creo que esto refleja cierto nivel de sesgo de confirmación. Cuando los científicos encuentran algo conservado entre las especies, lo celebran como un marcador ", dijo Megason. "Pero a menudo, todas las demás características no conservadas se ignoran. La información cuantitativa nos ayuda a superar algunos de estos sesgos ".
En otro sorprendente hallazgo, los equipos observaron que el proceso de diferenciación celular en distintos tipos de células, que comúnmente se cree que ocurre en una estructura similar a un árbol donde diferentes tipos de células se ramifican desde una célula ancestral común, también puede formar "bucles" como ramas.
Por ejemplo, la cresta neural, un grupo de células que dan lugar a diversos tipos de tejidos, incluyendo músculo liso, ciertas neuronas y hueso craneofacial, inicialmente surge de precursores neuronales y cutáneos, pero es bien conocido que genera células que parecen casi idénticas a los huesos y precursores de cartílago.
Los nuevos resultados sugieren que pueden ocurrir bucles similares en otras situaciones. Que las células en el mismo estado puedan tener historias de desarrollo muy diferentes sugiere que nuestra visión jerárquica del desarrollo como un "árbol" está demasiado simplificada, dijo Klein.
Los tres equipos también identificaron ciertas poblaciones celulares que existían en una especie de estado intermedio de "toma de decisiones". Schier y sus colegas descubrieron que, en ciertos puntos clave de la rama del desarrollo, las células parecían descender una trayectoria evolutiva pero luego cambiaban su destino a otra trayectoria.
Klein, Megason, Kirschner y sus colegas hicieron una observación relacionada de que, al principio del desarrollo, algunas células activaron dos programas de desarrollo distintos. Aunque esas células intermedias eventualmente adoptarán una identidad única, estos descubrimientos se suman a la imagen de cómo las células desarrollan su destino final y sugieren que puede haber factores más allá de los genes involucrados en la dirección del destino celular.
"Con células de múltiples líneas, tenemos que comenzar a preguntarnos si su destino final está siendo determinado por alguna fuerza selectiva o interacción con el medio ambiente, en lugar de solo programas genéticos", dijo Kirschner.
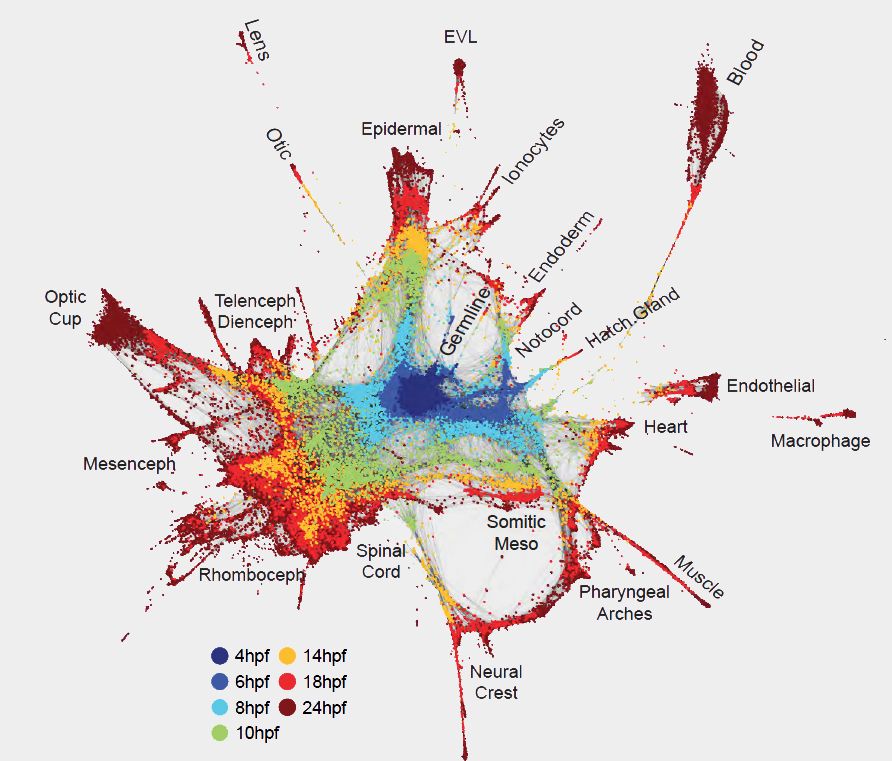 Una nueva visión del desarrollo embrionario que muestra más de 50,000 células organizadas por relaciones a lo largo del tiempo. El centro azul es el comienzo de la vida. Irradiando hacia afuera, surgen tipos celulares especializados. Imagen: Wagner et al.
Una nueva visión del desarrollo embrionario que muestra más de 50,000 células organizadas por relaciones a lo largo del tiempo. El centro azul es el comienzo de la vida. Irradiando hacia afuera, surgen tipos celulares especializados. Imagen: Wagner et al.
Fundación futura
Los nuevos conjuntos de datos generados y las nuevas herramientas y tecnologías desarrolladas como parte de estos estudios sientan las bases para un amplio espectro de exploración futura, según los autores.
Los biólogos del desarrollo pueden reunir más y más datos de calidad en muchas especies, seguir embriones en el tiempo y realizar cualquier cantidad de experimentos de perturbación, todo lo cual puede ayudar a mejorar nuestra comprensión de las reglas fundamentales de la biología y la enfermedad.
Estos recursos también pueden servir como un punto focal para la colaboración y la interacción, ya que la mayoría de los laboratorios no tienen la profundidad de la experiencia necesaria para explotar todos los datos y la información generada, señalaron los autores.
"Creo que estos estudios están creando un verdadero sentido de comunidad, con los investigadores planteando preguntas e interactuando entre sí de una manera que se remonta a épocas anteriores en el estudio de la embriología", dijo Kirschner.
Los tres estudios, dijo Schier, son un ejemplo de cómo la comunidad científica puede trabajar en preguntas complementarias para responder preguntas importantes en biología.
"En lugar de competir, nuestros grupos estuvieron en contacto regular durante los últimos dos años y coordinaron la publicación de nuestros estudios", dijo. "Y es muy bueno lo complementario de los tres documentos: cada uno destaca diferentes formas en que se pueden generar, analizar e interpretar conjuntos de datos tan complejos".
El próximo salto conceptual, sugieren los equipos, será comprender mejor cómo se toman las decisiones sobre el destino de las células.
"En este momento, tenemos una hoja de ruta, pero no nos dice cuáles son las señales", dijo Megason. "Lo que tenemos que hacer es descubrir las señales que dirigen las células hacia ciertos caminos, y cuáles son los mecanismos internos que permiten que las células tomen esas decisiones".
Independientemente del futuro, estos conjuntos de datos dejarán su marca.
"La belleza de trabajar en un organismo es que esto es todo", dijo Klein. "Diez, 20 años a partir de ahora, todavía podemos estar seguros de que el pez cebra y las ranas se desarrollarán de acuerdo con los mismos patrones".
Los tres equipos de investigación han puesto a disposición sus conjuntos de datos y herramientas como recursos interactivos y navegables en línea. Para Klein y sus colegas: pez cebra y Xenopus . Para Schier y sus colegas: pez cebra y URD .
Daniel Wagner es el autor principal y Sean Megason y Allon Klein son co-autores correspondientes del estudio " Cartografía unicelular de paisajes de expresión génica y linaje en el embrión de pez cebra ". Otros autores son Caleb Weinreb, Zach Collins y James Briggs. El estudio fue apoyado por los Institutos Nacionales de Salud (1K99GM121852, R01GM107733, R01DC015478), la Fundación Edward J. Mallinckrodt, Jr. y el Fondo Burroughs Wellcome.
James Briggs es el autor principal y Marc Kirschner y Allon Klein son co-autores correspondientes del estudio " La dinámica de la expresión génica en la embriogénesis de vertebrados en la resolución de células individuales ". Otros autores son Caleb Weinreb, Daniel Wagner, Sean Megason y Leonid Peshkin. El estudio fue apoyado por los Institutos Nacionales de Salud (R21HD087723), la Fundación Edward J. Mallinckrodt y el Fondo Burroughs Wellcome.
Jeffrey Farrell y Yiqun Wang son coautores principales y Aviv Regev y Alexander Schier son co-autores correspondientes del estudio " Reconstrucción unicelular de las trayectorias de desarrollo durante la embriogénesis del pez cebra ". Otros autores son Samantha Riesenfeld y Karthik Shekhar. El estudio fue apoyado por los Institutos Nacionales de Salud, el Centro de Descubrimiento Allen para el Rastreo de Linaje Celular, el Fondo Conmemorativo Jane Coffin Childs, el Fideicomiso Charles A. King, el Instituto Médico Howard Hughes y el Observatorio Celular Klarman.
Respuestas